2025.08.06
環境DNA分析のDNA配列情報から「真のDNA配列」を残して「より正確な遺伝的多様性評価」を可能にする新しい手法を開発

【本件のポイント】
- 環境DNA分析の配列データに記録された「真のDNA配列」に混じる「偽のDNA配列」を簡単かつ効果的に除去する新たな手法を開発し、インターネット上に公開
- 真のDNA配列がわかっているアユの環境DNAデータに本手法を適用し、その性能を評価。本手法が偽のDNA配列を高精度に選別・除去し、アユの種内遺伝的多様性をより正確に評価できることを実証
- 応用事例として、河川の魚類環境DNAを網羅的に調べた「メタバーコーディングデータ」に本手法を適用し、複数魚種のDNA配列を選別・抽出。種内の遺伝的多様性の知見があるアカザについて、既存の知見と一致するパターンを確認するとともに、未知のDNA配列を検出
- 今後、ますます増加する環境DNA調査に本手法を適用することで、種の多様性のみならず種内の遺伝的な多様性についても情報が蓄積され、生物保全や資源管理への活用が期待される
【研究成果の概要】
大妻女子大学家政学部の小関右介准教授、福井県里山里海湖研究所の武島弘彦研究員、龍谷大学生物多様性科学研究センターの山中裕樹教授らによる研究グループは、発展著しい環境DNA分析の技術のなかでも、次世代シーケンサーとよばれる装置を用いた分析により得られるDNA配列データに大量に混入する「ノイズ配列」あるいは「偽のDNA配列」を簡便かつ効果的に取り除き「真のDNA配列」を残す新たな配列フィルタリング手法を開発しました。
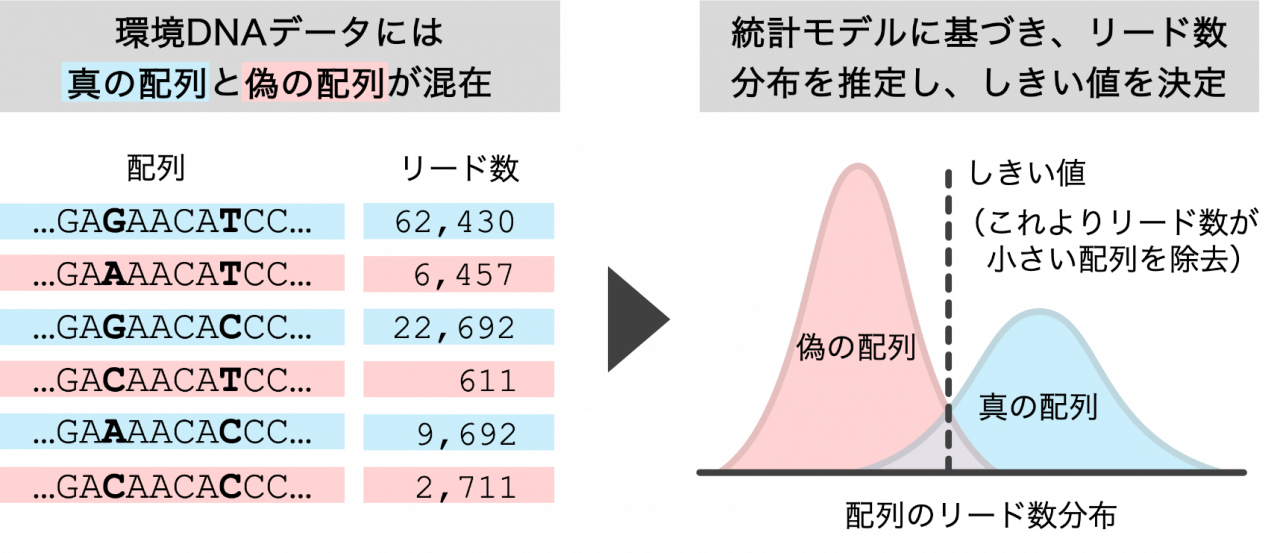
研究グループはまず、コンピューターシミュレーションを用いて、環境DNAデータに含まれる真のDNA配列と偽のDNA配列の配列ごとのリード数(読み取り量)の違いをはっきりと示し、その結果をもとに偽のDNA配列を選別・除去(フィルタリング)するしきい値を統計的に設定する手法を考案しました(図1)。この手法を広く分野の研究者に利用してもらうために、無償で利用できる統計解析ソフトウェア「R」の拡張パッケージ「gmmDenoise」を開発し、インターネット上に公開しました(https://github.com/YSKoseki/gmmDenoise)。
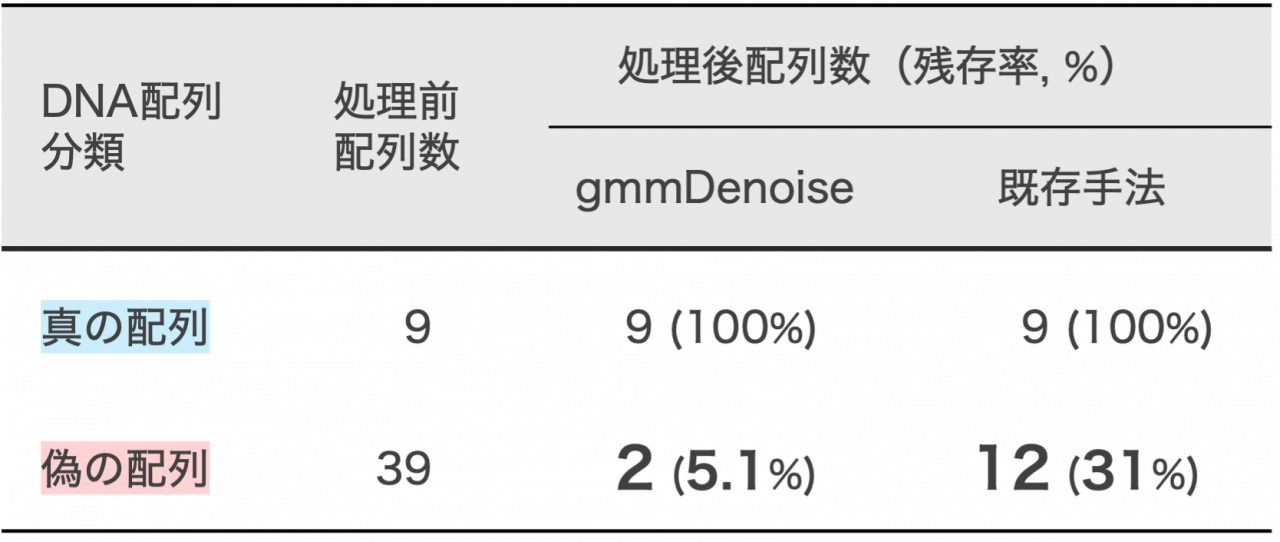
次に、本手法の性能を評価するために、真のDNA配列がわかっているアユの環境DNAデータについて、既存手法とフィルタリング成績を比較しました(表1)。この比較の結果、本手法は既存手法と比べて同等もしくはより高い精度で偽のDNA配列を選別・除去することが示されました。これらの結果は、本手法が優れた配列フィルタリング性能をもち、偽のDNA配列を含む環境DNAデータからの正確な遺伝的多様性の評価を可能にすることを示しています。
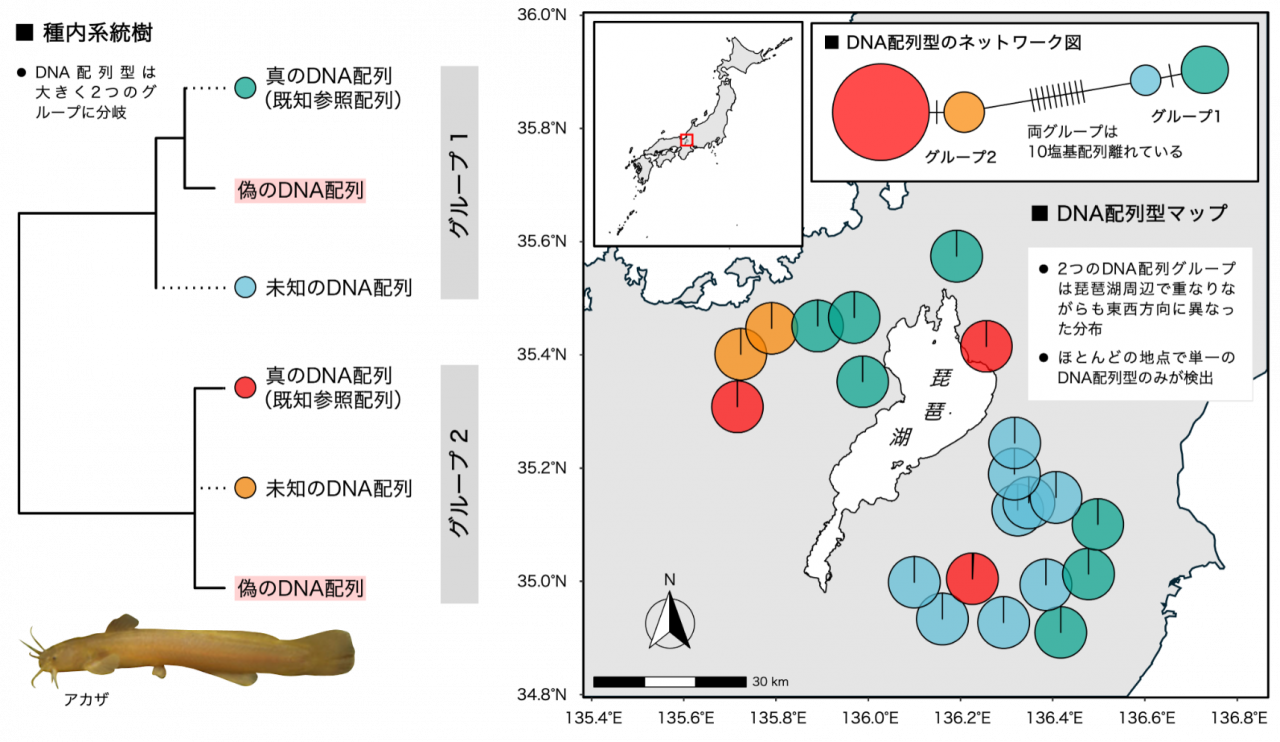
さらに、より実践的な解析事例として、河川の魚類群集の環境DNAを網羅的に解析した「メタバーコーディングデータ」に本手法を適用し、複数魚種のDNA配列を抽出しました。抽出したDNA配列のうち、先行研究から遺伝的多様性のパターンがわかっているアカザのDNA配列を詳しく分析したところ(図2)、既知の遺伝的多様性パターンとよく一致する結果が得られたうえ、これまで検出されなかった未知のDNA型も検出でき、本手法の有用性が実証されました。
表1.アユ飼育水から得た環境DNAデータにおけるgmmDenoise法と既存手法の配列フィルタリング処理の成績比較(結果の一部を抜粋)
図2.アカザのDNA配列型の分析結果(2つのDNA配列グループに分岐するアカザは、琵琶湖周辺で東西方向に異なる分布を示し、多くの地点で単一のDNA配列型のみを検出した)
【今後の展開】
現在、環境DNAデータの蓄積が急速に進んでいます。一方で、これまでの環境DNAデータの分析はおもに種の情報を得ることを目的としており、本手法のように、種がもつ遺伝的多様性の情報を客観的な基準で高精度に取得する方法はありませんでした。今後、さまざまな生物分類群や環境条件で得られた環境DNAデータへ本手法を適用することで、種の多様性のみならず種内の遺伝的多様性についても情報が蓄積され、生物多様性保全や資源管理への活用が期待されます。
【研究の背景】
水や土といった環境試料中に存在するDNAから生物情報を引き出す「環境DNA分析」は、直接的な生物の捕獲や観察を必要としない画期的な生物調査法としてその利用が急速に広がっています。そうした環境DNA分析の普及にともない、その用途も「この場所にどのような生物がいるのか」といった種の検出から、種の存続可能性や生物資源の健全性に重要な意味をもつ種内の遺伝的多様性の評価へと拡張されつつあります。しかし、環境DNA分析で得られるDNA配列データには、生物由来の真の配列に加えて、分析の過程(とくに塩基の並びを読み取る過程に先立ってDNA配列のコピーを量産するPCR過程)で生じるノイズ配列あるいは偽のDNA配列が相当数含まれることが知られており、この人為的に生じた偽のDNA配列をいかに除去して真のDNA配列を残すかが、環境DNAに基づく種内の遺伝的多様性評価の信頼性を高めるうえで大きな課題となっています。
この課題を解決するため、これまでに偽のDNA配列をフィルタリング(選別)して取り除く方法や類似する偽のDNA配列を真のDNA配列にまとめる手法がいくつか提案されてきましたが、いずれも効果が十分とはいえないうえ、フィルタリングの厳しさを指定する設定値を恣意的に決めているという問題がありました。そこで本研究は、偽のDNA配列の生成過程に関する理解に基づく新たな配列フィルタリング手法を開発し、その性能評価および実用性の検討を行いました。
【研究資金】
本研究は、JSPS科研費(JP21K12329、JP22K14908、JP25K02038)の助成を受けて実施されました。
【論文情報】
論文名:gmmDenoise: a new method and R package for high-confidence sequence
variant filtering in environmental DNA amplicon analysis
和 訳:gmmDenoise: 環境DNAアンプリコン解析における信頼性の高い配列フィルタ
リングのための新手法とRパッケージ
著者名:小関右介(責任著者)a,武島弘彦b c d,米田龍仁b,片柳海斗b,伊藤 玄e f,
山中裕樹e f
所 属:a大妻女子大学家政学部,b東海大学海洋学部,c福井県立大学海洋生物資源
学部,d福井県里山里海湖研究所(現在),e龍谷大学先端理工学部,
f龍谷大学生物多様性科学研究センター
掲載誌:Molecular Ecology Resources (John Wiley & Sons社)
公開日:2025年8月4日(早期オンライン公開)
URL:https://doi.org/10.1111/1755-0998.70023
【問い合わせ先】
<研究に関すること>
小関 右介(こせき ゆうすけ)
大妻女子大学家政学部ライフデザイン学科 准教授
Email: ykoseki@otsuma.ac.jp
<取材・報道に関すること>
・大妻女子大学 広報・入試センター 広報・募集グループ
TEL:03-5275-6011
E-mail: opr@ml.otsuma.ac.jp
・龍谷大学 研究部(生物多様性科学研究センター)
TEL:077-543-7746
E-mail: seibutsu-jimu@ad.ryukoku.ac.jp
URL: https://biodiversity.ryukoku.ac.jp/
・福井県里山里海湖研究所
TEL:0770-45-3580
E-mail: satoyama@pref.fukui.lg.jp