2021.07.07
様々なイネ系統の全ての遺伝子の応答を、気象情報から予測可能に 栽培地の環境を考慮したより高精度な育種を可能とする技術を開発
【本件のポイント】
- 野外圃場で生育期間を通じて様々なイネの系統の遺伝子発現を測定し、新たに開発したデータ解析手法によって、系統間の環境応答の差異を生み出すゲノム領域を同定
- 気象データとゲノムデータから任意の時点、系統の遺伝子発現を予測可能に
- 様々な栽培地や年度における遺伝子レベルからの応答予測が可能になることで、より高精度な育種が可能となり、食糧問題解決への一助へ
【本件の概要】
同じゲノム配列を持つ同一品種であっても、収量や品質は環境によって大きく変化します。これは、作物の遺伝子の発現量(※1)(どの遺伝子をどのくらい働かせるか)が環境によって変化するためです。その結果として地域間差、年次間差が生じます。我々のグループはこれまで、世界に先んじて、特定のイネ品種の遺伝子発現量の環境応答を予測する手法を確立してきました。しかしながら、別のイネ品種には応用できないという課題がありました。

図 1 本研究の概要
本研究では、新たに開発したデータ解析手法(edQTL解析)を用いて、イネ品種コシヒカリとタカナリに由来する多様な系統間における遺伝子発現量の環境応答の差異とゲノム多型との関連性を網羅的に調べ、1675遺伝子の環境応答の差異を生み出すゲノム領域を同定しました。さらに、同定したゲノム領域の情報をモデルに導入することで、任意の環境におけるコシヒカリ・タカナリ子孫系統の全遺伝子発現量の環境応答を高精度に予測できるようになりました。本研究で開発した遺伝子発現量予測手法は、他のイネ品種や他作物にも適用可能であり、栽培環境によって品種のパフォーマンスが異なるという育種における重要課題を解決する一助になると期待されます。
本研究は、龍谷大学農学部 永野惇准教授(慶應義塾大学先端生命科学研究所特任准教授)、龍谷大学食と農の総合研究所 鹿島誠特別研究員(現・青山学院大学理工学部化学・生命科学科助教)らを中心として、龍谷大学、東京農工大学、京都大学、青山学院大学、滋賀大学、済美高等学校、慶應義塾大学からなる研究チームによって実施されました。本研究は、科学技術振興機構 戦略的創造研究推進事業CREST「野外環境と超並列高度制御環境の統合モデリングによる頑健性限界の解明と応用(代表・永野惇)」プロジェクトの一環として実施されました。
タイトル:Genomic basis of transcriptome dynamics in rice under field conditions
著者:Makoto Kashima, Ryota L. Sakamoto, Hiroki Saito, Satoshi Ohkubo, Ayumi Tezuka,Ayumi Deguchi, Yoichi Hashida, Yuko Kurita, Koji Iwayama, Shunsuke Adachi and Atsushi J. Nagano
掲載誌:Plant and Cell Physiology, 2021, pcab088
【研究の背景】
同じゲノム配列を持つ作物(品種)であっても、栽培地や年度によって収量や品質にばらつきが生じてしまいます。これは、各々の作物が栽培される環境に応じて遺伝子の発現量を変化させているためだと考えられます。そのため、各栽培環境における収量や品質を予測するためには、遺伝子発現の環境応答を予測することが有効です。従来は、様々な品種を栽培し遺伝子発現量の違いとゲノム多型の違いを比較することで、eQTL(expression quantitative trait locus : 遺伝子の発現量に影響を与える座位)を同定していくアプローチがとられていました。しかし、一般的なeQTL解析では、測定した環境における遺伝子発現しか予測できないという大きな問題がありました(図2a)。
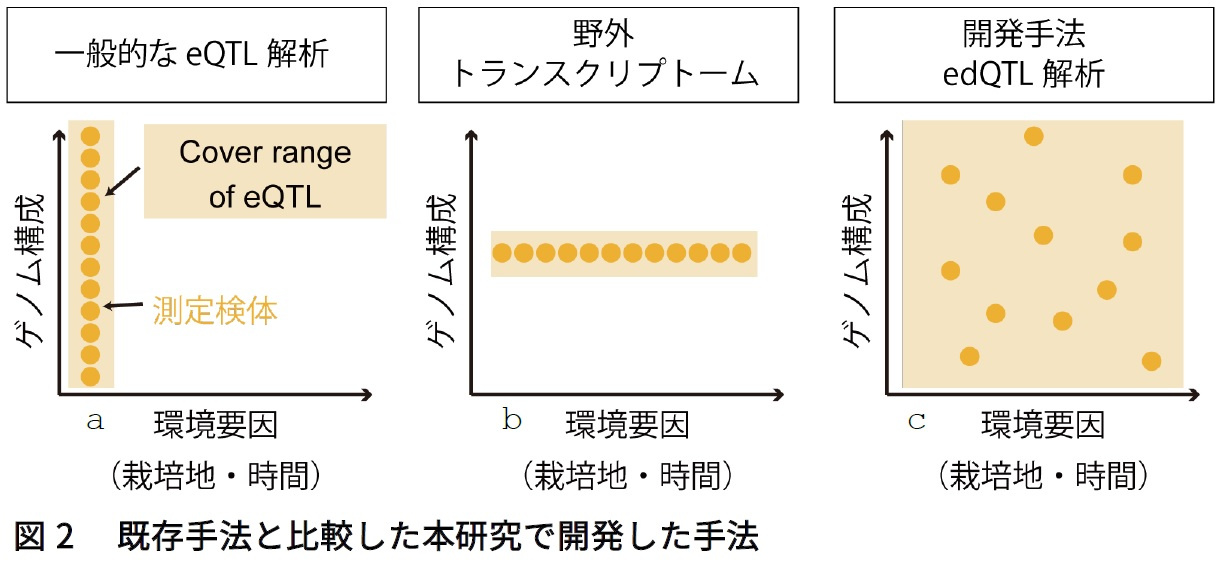
一方で、私たちの研究グループは、先行研究において開発した野外トランスクリプトームと呼ばれる手法を用いて、「日本晴」という品種に関し栽培地の気象情報と日時を指定することで遺伝子の発現量の環境応答を予測する統計モデルを作成しました。しかし、この環境応答予測モデルは「日本晴」専用であり、ゲノム配列が異なる別の系統に適用することはできませんでした(図2b)。育種の場面では、二つの系統を掛け合わせて、それぞれの系統の良いところを併せ持つ新品種を作成することが日常です。このような場合、親系統の環境応答を予測できても、子孫系統の環境応答は予測できないという問題があり、子孫系統の環境応答を予測する方法論の確立が求められていました(図2c)。
【研究の成果】
本研究では、日本で最も広く栽培されている品種「コシヒカリ」と多収品種である「タカナリ」に注目し、親系統及びその交配組み合わせによって生み出された子孫系統の環境応答を遺伝子発現レベルで予測する手法(遺伝子発現量予測手法)の開発を行いました。この子孫系統は、染色体断片置換系統(CSSL: Chromosome Segment Substitution Lines)と呼ばれるもので、ゲノムのほとんどは親系統であるコシヒカリあるいはタカナリと一致しますが、染色体の一部のみが他方の親系統に置換されており、染色体マーカーを調べることで、各ゲノム領域をコシヒカリ型かタカナリ型に区別することができます(図3)。
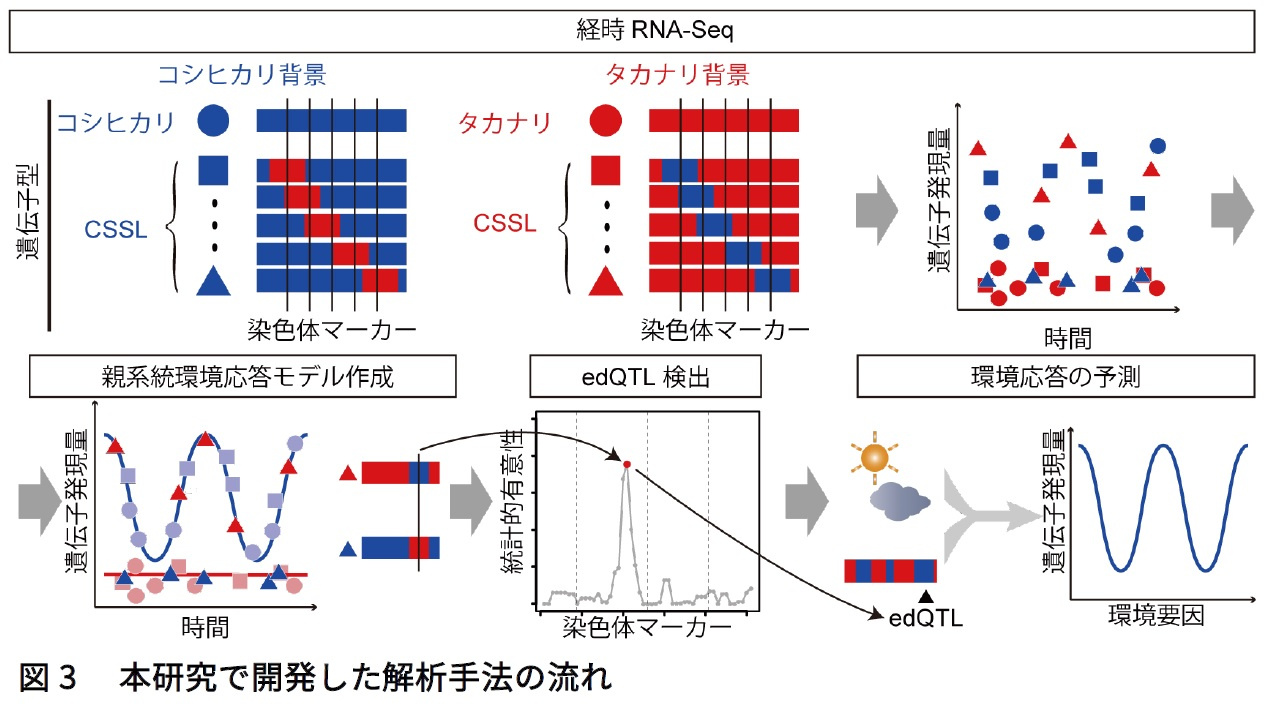
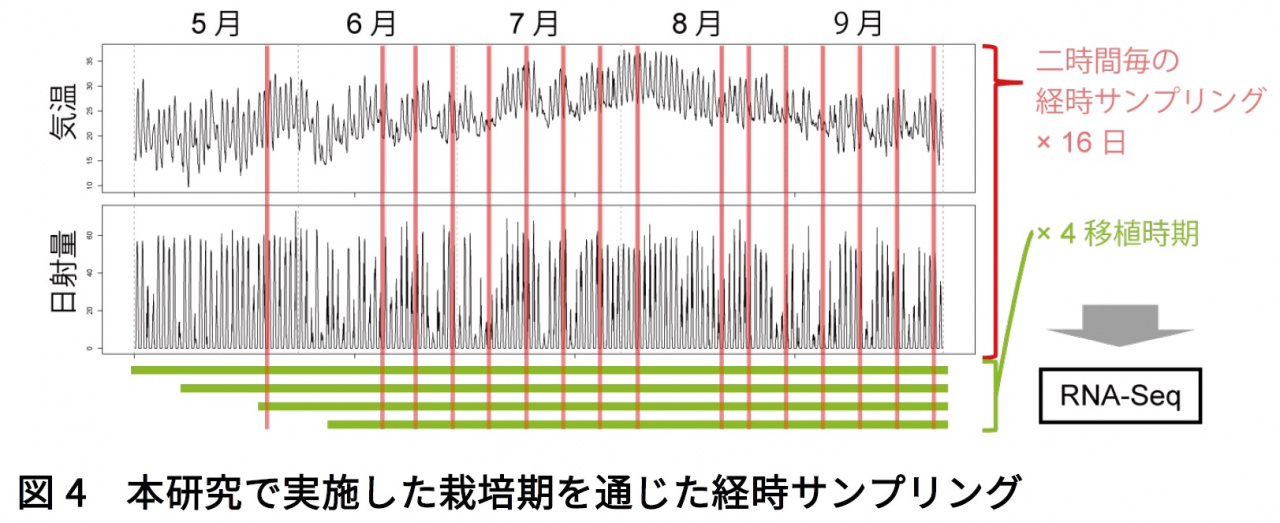
コシヒカリ、タカナリとCSSLの計80系統を旧京都大学大学院農学研究科附属農場(大阪府高槻市)で2015年に栽培し、2時間毎の24時間サンプリングを栽培期通じて複数回行いました(図4:赤線がサンプリング時期)。
その後、各個体の葉からRNAを抽出しRNA-Seq(※2)を行うことで、計845検体に関して全遺伝子の発現量を取得しました。
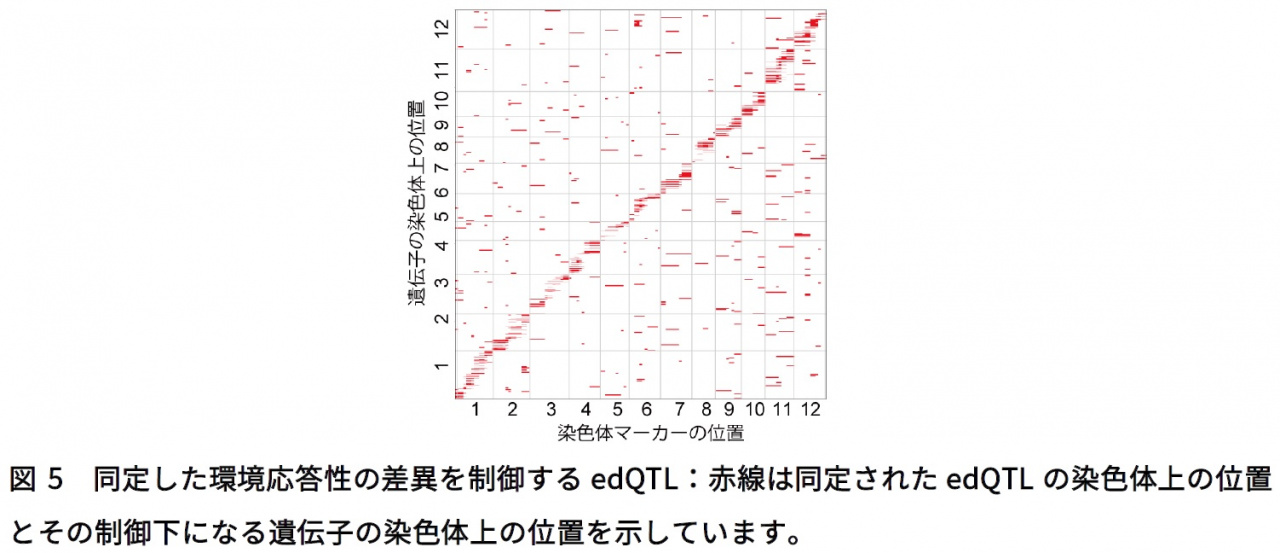
次に、アメダスから取得したサンプリング時の気温及び日射量と得られた全遺伝子発現量の情報をFIT(※3)と呼ばれる解析プログラムで解析することによって、任意の環境におけるコシヒカリ及びタカナリの遺伝子発現量を予測する環境応答モデルを作成し(図2c)、3696遺伝子がコシヒカリ・タカナリ間で異なる環境応答性を示すことを明らかにしました。さらに、「CSSLで親系統から置換された領域」と「環境応答モデルから予測した親系統(コシヒカリ、タカナリ)の各遺伝子の発現量と各サンプルの実測値のズレ」の関連性を統計的に検証することで、コシヒカリ・タカナリ間で異なる環境応答性を示す原因となるゲノム配列の多型を含む原因領域(edQTL: expression dynamics quantitative trait locus)を1675遺伝子に関して同定しました(図5)。
本研究において同定されたedQTLには、野外環境における作物の生育に重要な免疫応答の品種間差を生み出すゲノム領域も含まれていました。
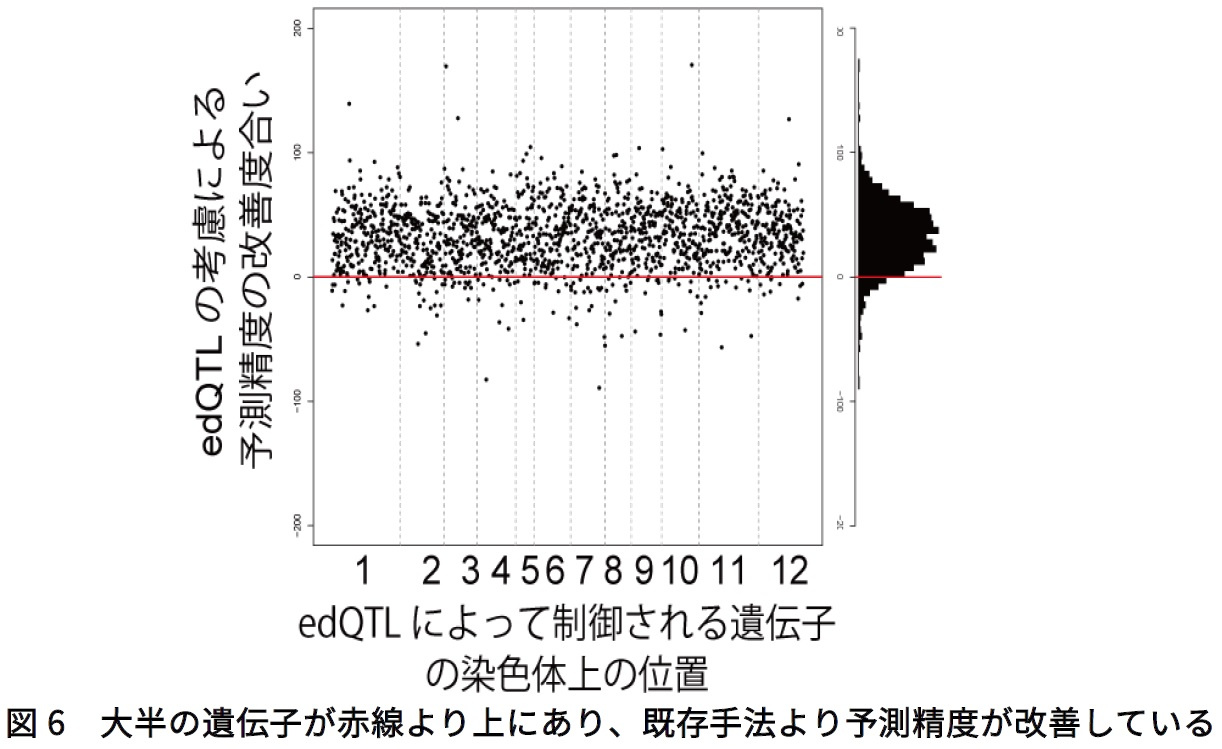
さらに、本研究では同定されたedQTLの情報をもとに実際に環境応答の予測が可能であるのかの検証を行いました。まず、異なる栽培地における環境応答の予測に同定したedQTLが有用であるのかの検証を行うため、翌年度(2016年)に京都大学大学院農学研究科附属農場(京都府木津川市)でCSSLを栽培、同様の経時RNA-Seqを行いました。edQTLを考慮せず背景となる親系統の環境応答モデルでの予測値、およびedQTLを考慮した予測値と、RNA-Seqによる実測値を比較すると、期待通りedQTLを考慮した方が遺伝子発現の予測精度が良く(図6)、異なる栽培環境における環境応答を予測できることが明らかになりました。
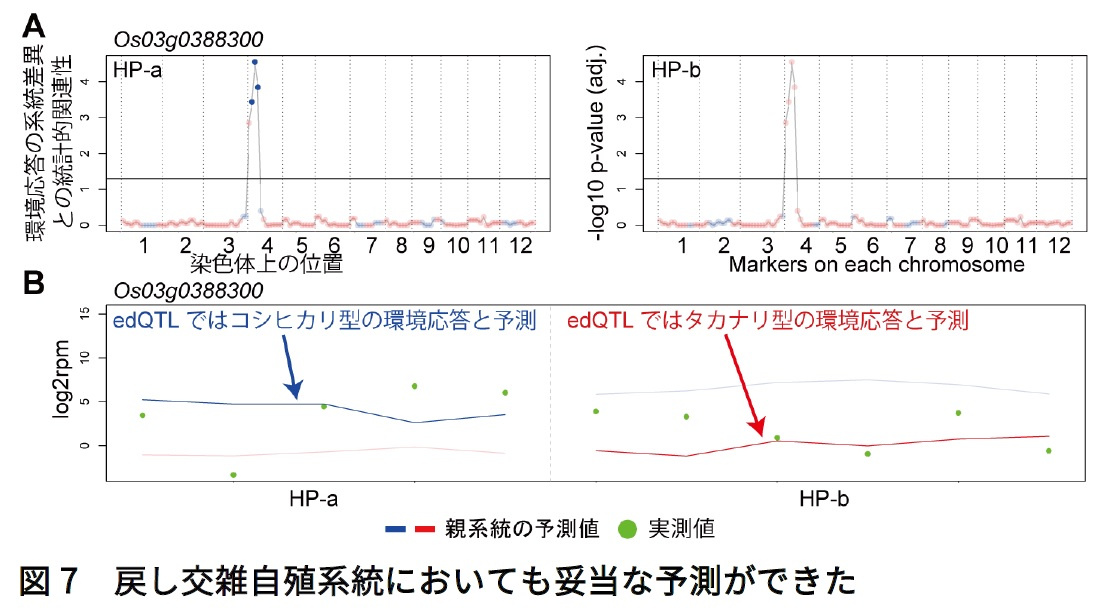
さらに、 edQTL同定に使用したCSSLよりも、よりゲノム領域の入れ替えが複雑な戻し交雑自殖系統(Backcross Inbred Lines:BIL)HP-aとHP-bに対して、環境応答の差異を正確に予測できることを確認しました。例えば、HP-a、 HP-b両系統はゲノムの大部分がタカナリ型ですが、Os03g0388300遺伝子の環境応答を制御するedQTLは、HP-aはコシヒカリ型であるのに対し、HP-bではタカナリ型です(図7A)。RNA-SeqによるOs03g0388300遺伝子の発現量の定量結果は、HP-aはコシヒカリ型環境応答モデルが、HP-bはタカナリ型環境応答モデルの予測結果が一致していました(図7B)。
【今後の期待】
本研究で確立した「親系統の遺伝子発現の環境応答の品種間差を生み出すedQTLの同定法」及び「edQTLを考慮した子孫系統の遺伝子発現の環境応答予測法」は、コシヒカリ・タカナリ以外のイネ品種だけでなく、その他の品種や作物にも適用可能なものです。現状の作物育種の課題として、栽培環境の差異から、必ずしも育種地と栽培地で収量や疫病耐性といった形質が一致しないという問題があります。形質の差異が主に遺伝子発現の差異に起因することを考慮すると、任意の環境応答における遺伝子発現の予測を可能とする本研究成果は、将来的には任意環境における形質予測を可能にし、任意の栽培地環境に適した品種の選定あるいは様々な環境に頑健な品種の作成に繋がる可能性を秘めています。
【注釈】
※1 遺伝子の発現量...mRNAの存在量のことを示しています。ゲノムには多数の遺伝子が存在しています。細胞内では、これらの遺伝子からmRNAと呼ばれるコピーが作られ、mRNAの情報が読み取られることでタンパク質が作られます。そのため、mRNA存在量≒遺伝子機能の発揮度合いといえます。遺伝子の発現量は、ゲノム配列の違いによって変化するため、環境応答の系統差を生み出します。
※2 RNA-Seq…次世代シーケンサーと呼ばれる超並列核酸配列解読装置を用い、サンプルに含まれるRNA(正確にはcDNA)を数えあげることで、数えられた頻度情報からRNAの存在量、すなわち遺伝子の発現量を網羅的に調べる手法です。
※3 FIT…オープンソースであるR言語で作成された、気象情報と経時的な遺伝子発現の測定結果から環境応答予測モデルを作成するプログラムです。ある系統に対して環境応答モデルを作成すれば、その系統の任意の栽培地・時点の遺伝子発現量を予測することが可能になります。
問い合わせ先:<研究内容>農学部 植物生命科学科 情報生物学研究室
准教授 永野 惇 Tel 077-599-5656
<担当部局>農学部教務課 森本 Tel 077-599-5601 Mail agr@ad.ryukoku.ac.jp