2025.08.08
フィルタリングで「真のDNA」を見抜く! 環境DNA分析データの精度を大幅に向上させる新手法を開発【生物多様性科学研究センター/先端理工学部】
環境DNA分析の配列データに記録された「真のDNA配列」に混じる「偽のDNA配列」を簡単かつ効果的に除去する新たな手法を開発し、インターネット上に公開
近年、生態系の監視や保全において「環境DNA分析」が注目されています。水や土などの環境中に含まれる生物由来のDNAから、生物の種類や分布を推定できる便利な技術ですが、実際には「ノイズ配列」または「偽のDNA配列」も大量に混ざり込んでしまいます。これは誤った生物種の検出や、多様性の評価ミスにつながる大きな問題でした。
このたび、大妻女子大学家政学部の小関右介准教授をはじめ、本学先端理工学部の山中裕樹教授(生物多様性科学研究センター長)、伊藤 玄博士研究員(生物多様性科学研究センター)、らが関わる共同研究チームは、環境DNA分析のDNA配列情報から「真のDNA配列」を残して「より正確な遺伝的多様性評価」を可能にする、新たな配列フィルタリング手法を考案しました。
また、この手法を広く分野の研究者に利用してもらうために、無償で利用できる統計解析ソフトウェア「R」の拡張パッケージ「gmmDenoise」を開発し、インターネット上に公開しました。
【新手法「gmmDenoise」の開発ポイント】
共同研究チームは、環境DNAメタバーコーディングで得られるDNA配列データに対し、「偽配列と真配列の読み取り数(リード数)分布の差」に注目。これをもとに、統計モデルを用いてノイズを判別し、偽配列を除去する“しきい値”を設定する手法を開発しました。
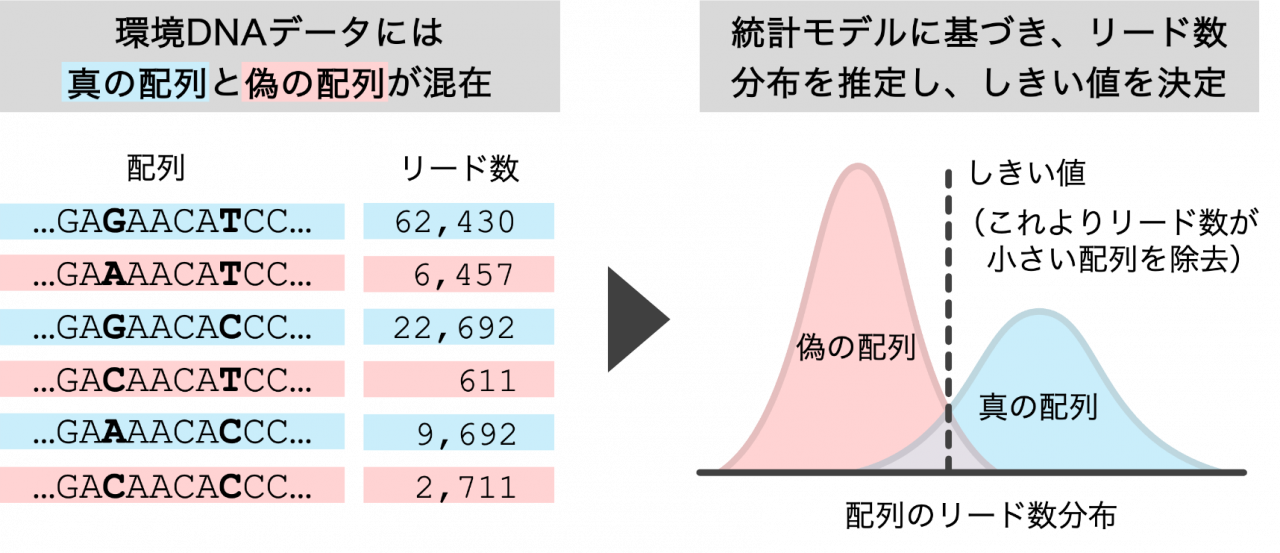
新たな手法による偽の配列の選別・除去の概念図
今回の研究成果について、Molecular Ecology Resources誌(John Wiley & Sons社)において公表しました。
→詳細はこちらのプレスリリース(2025年8月6日配信)を参照してください。
英文タイトル:gmmDenoise: A New Method and R Package for High-Confidence Sequence Variant Filtering in Environmental DNA Amplicon Analysis
タイトル和訳:gmmDenoise: 環境DNAアンプリコン解析における信頼性の高い配列フィルタリングのための新手法とRパッケージ
著者:小関右介(責任著者)a, 武島弘彦 b c d, 米田龍仁 b, 片柳海斗 b, 伊藤 玄 e f, 山中裕樹 e f
所属:a大妻女子大学家政学部, b 東海大学海洋学部, c福井県立大学海洋生物資源学部, d福井県里山里海湖研究所(現在), e 龍谷大学先端理工学部, f 龍谷大学 生物多様性科学研究センター
掲載誌:国際オンライン科学雑誌「Molecular Ecology Resources」(John Wiley & Sons社)
DOI:https://doi.org/10.1111/1755-0998.70023
掲載日:2025年8月4日(早期オンライン公開)
研究資金:JSPS科研費(JP21K12329、JP22K14908、JP25K02038)
今回の研究成果に関して、当センターの研究メンバーのコメントを紹介します。
◉山中裕樹教授(本学先端理工学部/生物多様性科学研究センター長)
「環境DNA分析では得られたDNA配列情報が『真の答えかどうか』を厳密に確認することができない場面が多いです。その信頼性を担保するのに、今回のツールは役に立てると思います。同種であっても地域的にわずかに異なる遺伝的背景を持っている生物について詳細に分布域を明らかにするなど、保全の場面で有効だと考えています。」

山中裕樹教授(本学先端理工学部/生物多様性科学研究センター長)

伊藤 玄博士研究員(生物多様性科学研究センター)
◉伊藤 玄博士研究員(生物多様性科学研究センター)
「今回の研究成果は、私がこれまでに取り組んできた、生物の地理的分布と遺伝的多様性の違いを明らかにする研究分野へ環境DNAを適用するために、欠かせない技術になると思います。世界中の研究者に活用されることを願っています。」